基于酶切的簡化基因組測序(Restriction-site Associated DNA Sequence, RAD-Seq)是對與限制性核酸內切酶識別位點相關的DNA進行高通量測序,可大幅降低基因組的復雜度,降低建庫和測序成本,操作簡便,同時不受參考基因組的限制,可快速鑒定出高密度的SNP位點,實現遺傳進化分析及重要性狀候選基因的預測。RAD-Seq尤其適合于大樣本量的研究,可以為利用全基因組重測序技術做深度信息挖掘奠定堅實的基礎。RAD-Seq技術可廣泛應用于變異檢測、遺傳圖譜構建、功能基因挖掘、群體進化等研究,具有重大的科研和產業價值。
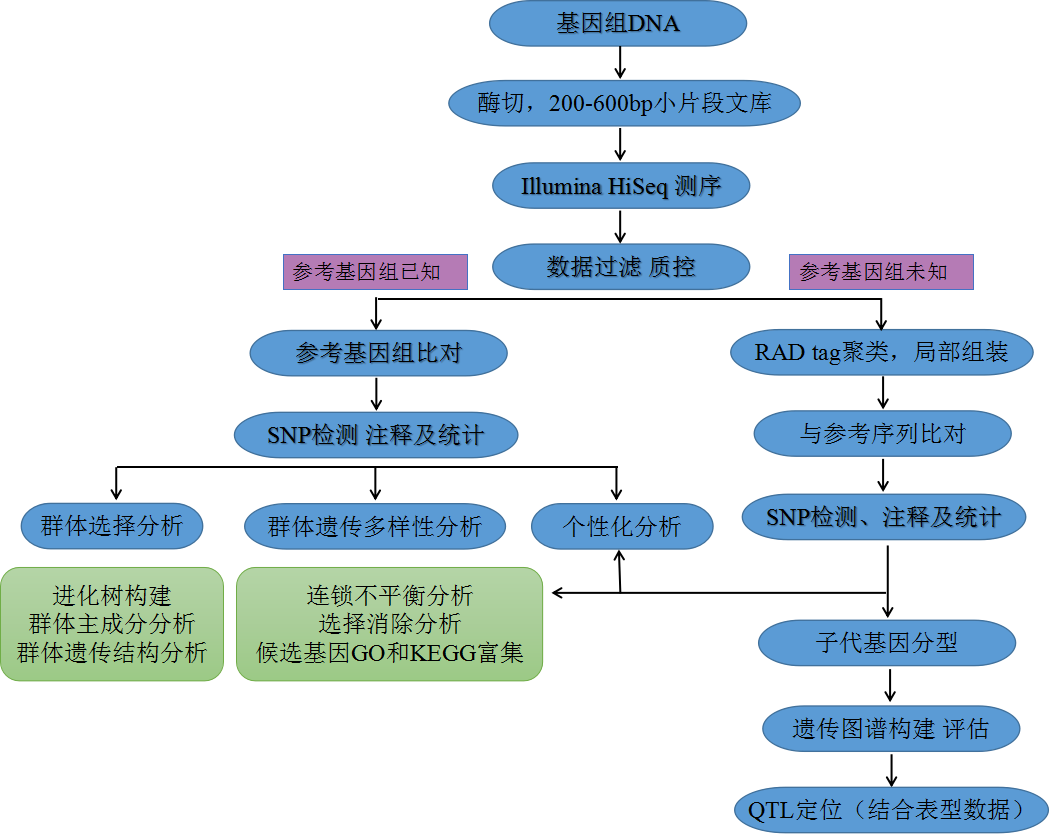
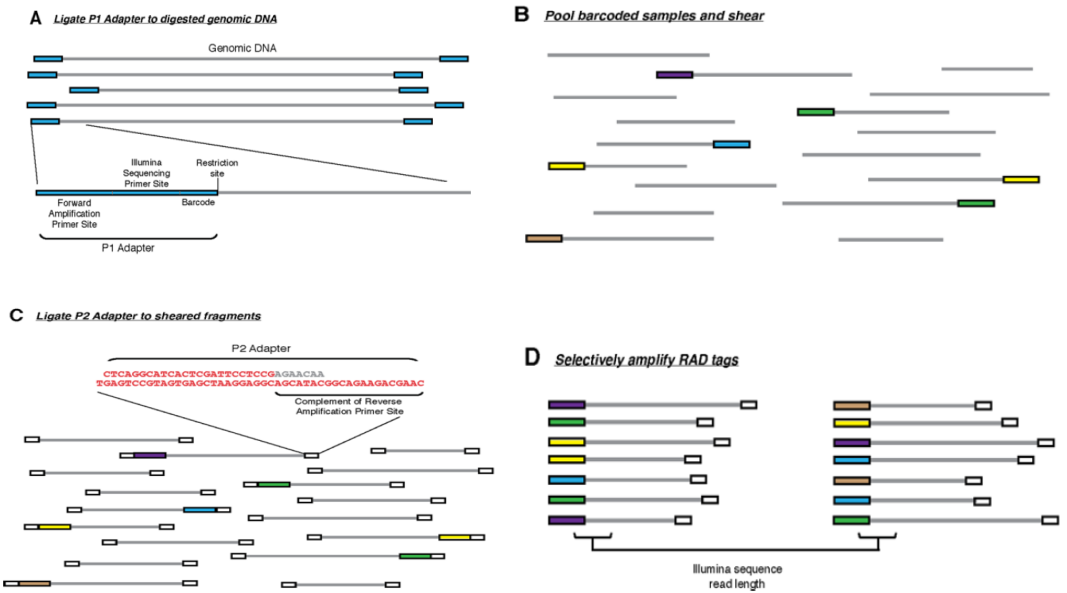
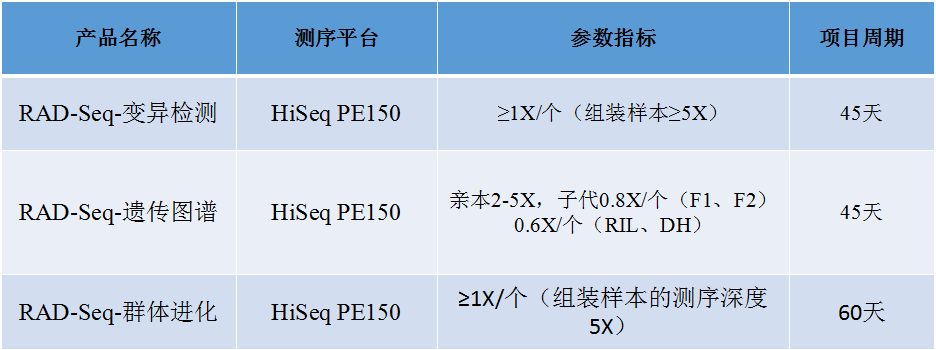
一、變異檢測:
1. DNA樣品量≥ 3 ug;
2. EcoRI(GAATTC)酶切;
3. 推薦測序深度≥ 1X/個(組裝樣本的測序深度≥ 5X)。
二、遺傳圖譜:
1. DNA樣品量≥ 3 ug;
2. 測序深度:親本2-5X,子代0.8X/個(F1、F2等臨時性群體),0.6X/個(RIL、DH等永久性群體);
3. 適用范圍:單倍體或者二倍體物種,所有作圖群體(F1、F2;RIL、DH等),群體大小在100個以上。
三、群體進化:
1. DNA樣品量≥ 3 ug;
2. 測序深度:≥ 1X/個(組裝樣本的測序深度5X)
3. 適用范圍:單倍體或者二倍體物種中不同亞群,各亞群間劃分明顯,同一亞群內的個體有一定代表性,每個亞群選取10個樣本左右(動物≥ 10個,植物≥ 15個),總體不少于30個樣本。
四、樣品細則:
1. DNA樣品:請提供濃度>100 ng/μl,總量>3μg的DNA,OD 260/280在1.8~2.0之間,并確保DNA無降解,電泳檢測無明顯RNA條帶,基因組條帶清晰、完整,主帶應在100kb以上。若樣品中有多糖、糖蛋白的殘留,對打斷DNA樣品帶來非常大的困難,且很難去除,因此特別要求所提供的樣品不要有多糖或糖蛋白污染。
2. 植物樣品:選取植物幼嫩組織,每個樣品重量>500 mg,干冰或者液氮保存寄送。
3. 動物樣品:要求新鮮動物組織,避免脂肪組織,每個樣品重量>50 mg。對于一般物種應挑選肝臟、腎臟、血液等組織取樣,對于珍貴物種請提供耳樣、毛發(帶毛根)等脂肪含量較少的組織進行取樣。為了減少個體差異對后續拼接產生的影響,盡量從同一個個體中取樣。若物種體積較小,從一個個體中提取的DNA量不能滿足測序實驗所需,在保證量的前提下,應盡量減少采樣個體的數量。提供組織樣品應>50 mg,盡量提供較多量,不同的物種DNA提取產量有差異。
4. 樣品運輸:送樣管務必標清樣品編號,管口使用Parafilm膜密封,樣品請用干冰或者冰袋運輸。干冰運輸時間不要超過72小時;冰袋運輸,時間最好不要超過24小時。樣品保存期間切忌反復凍融。
1. Q:高密度SNPs標記開發時要考慮哪些因素?
A:高密度SNPs標記開發的基本條件和設計理念是所獲得的片段盡量在基因組上分布均勻,通過測定少量代表全基因組信息的序列獲得成千上萬個SNPs標記。首先要利用生物信息學方法對目標物種參考基因組(或已知BAC序列)進行系統分析,根據基因組GC含量、重復序列情況和基因特點等信息,選擇相應的限制性酶以及測序文庫類型,以保證分子標記的密度、均勻性、效率滿足遺傳分析和分子育種的需要。
2. Q:RAD-seq的適用范圍?
A:可用于鑒定任何物種(有無參考基因組數據均可)的遺傳圖譜、多態(親緣地理分析)、關聯、QTL定位等分析。RAD-Seq技術可廣泛應用于變異檢測、遺傳圖譜構建、功能基因挖掘、群體進化等研究。
3. Q:用RAD-seq研究群體進化的優勢是什么?
A:一般情況下,群體進化研究涉及較大的樣本量,RAD-seq可大幅降低基因組的復雜度,降低建庫和測序成本,操作簡便。另外,它不受參考基因組的限制,研究物種范圍更廣泛。RAD-Seq尤其適合于大樣本量的研究,可以為利用全基因組重測序技術做深度信息挖掘奠定堅實的基礎。
4. Q:有參和無參的區別是什么?
A:對于發現突變信息而言,有參考基因組進行序列比對和變異檢測的效率和準確率更高。而且,可以定位受選擇的基因,研究群體的連鎖不平衡現象。
